Методические рекомендации MP 3.1.0272-22 "Молекулярно-генетический мониторинг штаммов возбудителя новой коронавирусной инфекции" (утв. Федеральной службой по надзору в сфере защиты прав потребителей и благополучия человека 10 января 2022 г.)
Методические рекомендации MP 3.1.0272-22
"Молекулярно-генетический мониторинг штаммов возбудителя новой коронавирусной инфекции"
(утв. Федеральной службой по надзору в сфере защиты прав потребителей и благополучия человека 10 января 2022 г.)
I. Область применения
1.1. Настоящие методические рекомендации информируют об основных принципах организации и проведения молекулярно-генетического мониторинга штаммов возбудителя новой коронавирусной инфекции, циркулирующих на территории Российской Федерации.
1.2. Настоящие методические рекомендации предназначены для специалистов органов и организаций Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека, а также иных организаций, участвующих в проведении данного мониторинга.
1.3. Перечень научных организаций, противочумных учреждений Роспотребнадзора, осуществляющих полногеномное и фрагментное (мультилокусное) секвенирование материала от больных новой коронавирусной инфекцией, доставленного территориальными органами и организациями Роспотребнадзора в субъектах Российской Федерации, иными организациями, представлен в приложении 1 к настоящим методическим рекомендациям.
II. Критерии забора материала для определения геновариантов VOC SARS-COV-2 с помощью секвенирования
2.1. Приоритетные критерии для забора материала для секвенирования:
- от лиц, прибывших из-за рубежа;
- от контактных с прибывшими из-за рубежа;
- от контактных с лицами, у которых выявлен штамм SARS-CoV-2 "вызывающий озабоченность" (VOC);
- от лиц с подозрением на повторное инфицирование SARS-CoV-2;
- от лиц, инфицированных (с признаками и без признаков заболевания), спустя 30 и более суток после вакцинации;
- от больных с устойчивостью к лечению препаратами на основе специфических антител;
- от всех лиц с нетипичным течением заболевания (длительная вирусемия более 21 дня, наличие симптомов со стороны желудочно-кишечного тракта, нервной системы и др.) без сопутствующей патологии;
- от лиц из эпидемических очагов (в т.ч. семейных);
- от детей и подростов до 17 лет с клиническим признаками заболевания.
С учетом складывающейся эпидемиологической ситуации в субъекте Российской Федерации критерии для забора материала для секвенирования могут быть изменены.
2.2. Дополнительно целесообразно обеспечить забор материала с "потока" - от амбулаторных и стационарных пациентов.
2.3. Общий объем направленных на секвенирование проб из субъектов Российской Федерации - не менее 10% от всех положительных исследований (ПЦР) в неделю.
2.4. Доставка биоматериала осуществляется не менее двух раз в неделю по согласованию с лабораториями, проводящими секвенирование.
2.5. Требования к отбору материала.
2.5.1. В случае положительного результата ПЦР отбирается носоглоточный мазок в транспортной среде, который затем отправляется в соответствующую лабораторию. Допустимые для использования типы транспортных сред определяются строго по согласованию с организацией, осуществляющей секвенирование.
2.5.2. Лаборатории, проводящие секвенирование, самостоятельно определяют пригодность проб для проведения секвенирования.
2.5.3. Образцы необходимо подвергать глубокой заморозке и хранить до передачи в лабораторию в условиях, не допускающих их повторной разморозки, обеспечивая холодовую цепочку при транспортировке материала; при пересылке в течение 24 часов и менее от момента отбора материала образцы допускается перевозить при +4°С, не замораживая.
2.5.4. Для отбора респираторных мазков используются пробирки для взятия респираторных мазков, содержащих транспортную среду. Допускается использование криопробирок объемом 1,5-2 мл с внешней резьбой и завинчивающимися крышками с уплотнительным кольцом, исключающим протекание биологической жидкости, в которые внесена транспортная среда в объёме 0,6 мл. Недопустимо использовать другой тип пробирок. Пробирки с материалом должны иметь четкую несмываемую маркировку.
III. Требования к сопроводительной информации о предоставляемом материале
3.1. Сопроводительная информация должна предоставляться организациям, осуществляющим секвенирование, через заполнение онлайн-формы в соответствии с инструкцией, представленной в приложение 2 "Инструкция по регистрации образца".
3.2. После загрузки обязательной информации образцу выдаётся уникальный идентификационный номер и формируется сопроводительный лист. Сопроводительный лист распечатывается и вкладывается в посылку, содержащую контейнер с образцами.
3.3 Доступ к заполнению онлайн-формы осуществляется ответственным лицом организации, направляющей образцы на исследование. Для входа в систему используется логин и пароль, предоставляемые по запросу в ФБУН "ЦНИИ Эпидемиологии" Роспотребнадзора. Для получения доступа в систему требуется предоставить информацию в ФБУН "ЦНИИ Эпидемиологии" Роспотребнадзора об ответственном сотруднике (название организации, должность, ФИО, электронная почта, мобильный телефон, заполненный документ приложение 3 "Обязательство о неразглашении конфиденциальной информации").
3.4. Сопроводительная информация должна содержать перечисленные ниже данные об образце (обязательные поля выделены жирным шрифтом и отмечены "*"):
*Название образца (без пробелов, латиница, цифры, нижнее подчеркивание, дефис)
- Тип биоматериала - например, мазок из носо- или ротоглотки, аутопсия и т.п.
- *Место забора материала (в формате Страна/Регион/Город)
- *Дата забора материала в формате (ГГГГ или ГГГГ-ММ или ГГГГ-ММ-ДД)
- *ID образца (идентификационный номер образца из системы report.gsen.ru форма 970)
- *Субъект РФ (в формате Регион/Город)
- Пол пациента - М/Ж
- Возраст пациента - полных лет или неизвестен
- Социальный статус (род занятий/место работы)
- *Ct на ПЦР-тесте
- *Тест-система
- *Завозной случай - ДА/НЕТ
- *Если завозной случай, то дата прибытия в формате (ГГГГ или ГГГГ-ММ или ГГГГ-ММ-ДД)
- *Если завозной случай, то откуда прибыл (Страна прибытия/населенный пункт)
- Дата заболевания (ГГГГ или ГГГГ-ММ или ГГГГ-ММ-ДД)
- Диагноз по МКБ-10
- Клиническая форма заболевания - бессимптомно/ОРВИ/ВП/неизвестно
- Симптомы
- Исход заболевания - болеет/выздоровел/умер/неизвестно
- Госпитализация - - ДА/НЕТ/неизвестно
- *Подозрение на повторное инфицирование SARS-CoV-2 - ДА/НЕТ/неизвестно
- *Вакцинация - Привит/Не привит/Не известно
- *Название вакцины (если привит)
- Дата вакцинации (если привит)
- Кратность вакцинации - v1/v2
- Количество контактных лиц
- Из них выявлено лиц с COVID-19
- Если образцы от контактных лиц с COVID-19 поступили на секвенирование (указать идентификационные номера образцов из системы report.gsen.ru форма 970, если известно)
- Предположительный источник инфекции (если установлен)
- Комментарии (свободное поле)
IV. Требования к транспортированию материала
4.1. Материал помещается в герметичный, предварительно охлажденный до температуры хранения образцов металлический контейнер (термос с широким горлом), обеспечивающий сохранность терморежима. Допускается поместить в контейнер охлаждающие элементы. Во избежание аварий недопустимо помещать внутрь металлического контейнера (термоса) сухой лед или жидкий азот.
4.2. К наружной поверхности прикрепляют этикетку, содержащую информацию об организации, направившей материал, идентификационные данные материала с перечислением образцов, дату упаковки.
4.3. Маркированный контейнер помещают в термоизолирующую пенопластовую коробку (термоконтейнеры, сумки-термосы), содержащую замороженные хладоэлементы и/или сухой лед в количестве, достаточном для обеспечения непрерывной холодовой цепи до места доставки.
4.4. К наружной стенке коробки прикрепляют этикетку с указанием вида материала, условий транспортирования, названия пунктов назначения, получателя и отправителя с контактными данными. Не допускается указание на этикетке снаружи упаковки любых данных, помимо вышеуказанных.
4.5. Перевозка образцов должна осуществляться в соответствии с требованиями санитарного законодательства по отношению к микроорганизмам II группы патогенности.
4.6. Биоматериал необходимо доставлять в научные организации Роспотребнадзора в течение не более 5 суток от момента забора.
V. Алгоритм дифференциации генетических вариантов вируса SARS-COV-2
5.1. Актуальный список вариантов, подлежащих исследованию, с указанием искомых мутаций, а также их распределения по амплифицируемым фрагментам из протоколов Университета Женевы и ARTIC v.3 или других приводится в техническом бюллетене, обновляемом 1 и 15 числа каждого месяца на портале https://genome.crie.ru/.
В случае оперативной необходимости бюллетень может быть дополнительно актуализирован.
Информация об актуализации данных поступает в виде информационной рассылки с портала https://genome.crie.ru/ в научно-исследовательские организации Роспотребнадзора, осуществляющие проведение секвенирования (приложение 4).
5.2. Для дифференциации геновариантов вируса SARS-CoV-2 используются методы (1) фрагментного секвенирования по Сэнгеру отдельных локусов гена, кодирующего S-белок, (2) секвенирования полной последовательности S-гена или (3) секвенирование полного генома вируса SARS-CoV-2.
5.3. При применении методов секвенирования последовательно проводится (1) амплификация необходимых фрагментов генома SARS-CoV-2, определения наличия продуктов амплификации, (2) секвенирование пригодных образцов выбранным методом, из числа перечисленных ниже, (3) выдача заключения о вероятной принадлежности к искомому геноварианту.
5.4. Сроки проведения оценки качества (амплификация необходимых фрагментов генома SARS-CoV-2, определение наличия продуктов амплификации) - не более 3 суток от момента поставки образцов (96 или менее).
Сроки проведения фрагментного секвенирования пригодных образцов (любым выбранным протоколом) - не более 4 дней от момента получения заключения о качестве.
Сроки проведения секвенирования гена S-белка (любым выбранным протоколом) - не более 7 дней от момента получения заключения о качестве.
Сроки проведения полногеномного секвенирования пригодных образцов (любым выбранным протоколом) - не более 12 дней от момента получения заключения о качестве.
Сроки выдачи заключения о вероятной принадлежности вируса к одному из искомых вариантов - образца не более 1 дня от момента завершения секвенирования.
5.5. Организациям, участвующим в секвенировании, рекомендуется обращать внимание на новые возникающие геноварианты и при закреплении таких в популяции сообщать о них, высылая письмо с описанием геноварианта и рекомендациями по выявлению по адресу crie@pcr.ru".
5.6. Инструкция по работе с программой genome.crie.ru приведена в приложении 5 к настоящим методическим рекомендациям.
Приложение 1
к MP 3.1.0272-22
Перечень
научных организаций, противочумных учреждений Роспотребнадзора, осуществляющих секвенирование
| Перечень учреждений, осуществляющих секвенирование | Субъекты | |
|---|---|---|
| Фрагментное (мультилокусное) | Полногеномное | |
| ФБУН "Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора" | ФБУН "Центральный научно-исследовательский институт эпидемиологии Роспотребнадзора" | Владимирская, Ивановская, Костромская, Рязанская, Смоленская, Тверская, Липецкая, Тамбовская, Ярославская области Москва Магаданская область, Чукотский автономный округ, Камчатский край |
| ФБУН "Государственный научный центр прикладной микробиологии и биотехнологии" Роспотребнадзора | ФБУН "Государственный научный центр прикладной микробиологии и биотехнологии" Роспотребнадзора | Калужская, Московская, Орловская, Тульская Белгородская, Воронежская Брянская, Курская области |
| ФБУН "Санкт-Петербургский научно-исследовательский институт эпидемиологии и микробиологии имени Пастера" Роспотребнадзора | ФБУН "Санкт-Петербургский научно-исследовательский институт эпидемиологии и микробиологии имени Пастера" Роспотребнадзора | Республики Карелия, Коми, Ненецкий автономный округ, Архангельская, Вологодская, Ленинградская, Новгородская, Псковская Мурманская Калининградская области г. Санкт-Петербург |
| ФКУЗ "Российский научно-исследовательский противочумный институт "Микроб" Роспотребнадзора | ФКУЗ "Российский научно-исследовательский противочумный институт "Микроб" Роспотребнадзора | Республики Башкортостан Удмуртская Татарстан Самарская Пензенская, Саратовская, Ульяновская Оренбургская области |
| ФБУН "Нижегородский научно-исследовательский институт эпидемиологии и микробиологии имени академика И.Н. Блохиной" Роспотребнадзора | ФКУЗ "Российский научно-исследовательский противочумный институт "Микроб" | Республики Мордовия Марий Эл Чувашская Нижегородская Кировская области, Пермский край |
| ФБУН "Тюменский научно-исследовательский институт краевой инфекционной патологии" Роспотребнадзора | ФБУН "Государственный научный центр вирусологии и биотехнологии "Вектор" Роспотребнадзора | Челябинская Курганская Свердловская Тюменская области, Ханты-Мансийский Ямало-Ненецкий автономные округа |
| ФКУЗ "Волгоградский научно-исследовательский противочумный институт" Роспотребнадзора | ФКУЗ "Волгоградский научно-исследовательский противочумный институт" Роспотребнадзора | Волгоградская Астраханская области Республика Калмыкия |
| ФКУЗ "Ставропольский научно-исследовательский противочумный институт" Роспотребнадзора | ФКУЗ "Ставропольский научно-исследовательский противочумный институт" Роспотребнадзора | Краснодарский край Республика Адыгея |
| ФБУН "Ростовский научно-исследовательский институт микробиологии и паразитологии" Роспотребнадзора | ФКУЗ "Ростовский научно-исследовательский противочумный институт" Роспотребнадзора | Ростовская область Крым, г. Севастополь |
| ФКУЗ "Ставропольский научно-исследовательский противочумный институт" Роспотребнадзора | ФКУЗ "Ставропольский научно-исследовательский противочумный институт" Роспотребнадзора | Республики Северная Осетия - Алания, Ингушская, Чеченская, Дагестан Кабардино-Балкарская, Карачаево-Черкесская, Ставропольский край |
| ФБУН "Омский научно-исследовательский институт природно-очаговых инфекций" Роспотребнадзора | "Государственный научный центр вирусологии и биотехнологии "Вектор" Роспотребнадзора | Омская, Томская, Кемеровская области |
| ФКУЗ "Иркутский научно-исследовательский противочумный институт Сибири и Дальнего Востока" Роспотребнадзора | ФКУЗ "Иркутский научно-исследовательский противочумный институт Сибири и Дальнего Востока" Роспотребнадзора | Иркутская область Республика Бурятия, Забайкальский край Республика Хакасия, Красноярский край Республика Алтай, Алтайский край Республика Тыва |
| ФБУН "Государственный научный центр вирусологии и биотехнологии "Вектор" Роспотребнадзора | "Государственный научный центр вирусологии и биотехнологии "Вектор" Роспотребнадзора | Новосибирская область |
| ФБУН "Хабаровский научно-исследовательский институт эпидемиологии и микробиологии" Роспотребнадзора | "Государственный научный центр вирусологии и биотехнологии "Вектор" Роспотребнадзора | Республика Саха (Якутия), Хабаровский край, Приморский край, Еврейская автономная область, Сахалинская, Амурская область |
| ФБУН "Научно-исследовательский институт дезинфектологии" Роспотребнадзора | Коммерческие организации осуществляющие молекулярно-генетические исследования (ПЦР) | |
Приложение 2
к MP 3.1.0272-22
Требования к файлам, содержащим информацию о нуклеотидной последовательности
Каждый геном SARS-CoV-2 должен быть представлен одной нуклеотидной последовательностью в форматах "*.fasta", "*.fas", "*.fa".
Требования к наименованию последовательности:
- должно содержать латинские буквы, цифры, нижнее подчёркивание, не должно содержать пробелов
- должно быть полностью идентично названию образца, введенному в форму для представления метаданных.
Требования к буквенным обозначениям нуклеотидов в файлах:
- в случае наличия не покрытых (неотсеквенированных) областей в геноме, они должны быть замаскированы последовательностью, состоящей из букв NNN;
- в случае наличия вырожденных нуклеотидных позиций они обозначаются вырожденным кодом, согласно номенклатуре IUРАС (например R, для обозначения A/G и т.п.)".
Приложение 3
к МР 3.1.0272-22
Обязательство о неразглашении конфиденциальной информации
Я, _______________________________________________________________,
(фамилия имя отчество)
выполняя свои должностные обязанности ___________________________________
(наименование должности)
_________________________________________________________________________
в НАЗВАНИЕ ОРГАНИЗАЦИИ и являясь непосредственным участником проекта, реализуемым в соответствии с Постановлением Правительства Российской Федерации от 23 марта 2021 N 448 и приказом Роспотребнадзора от 19.02.2021 N 56 "О совершенствовании молекулярно-генетического мониторинга штаммов возбудителя новой коронавирусной инфекции" (далее - Проект), получаю доступ к конфиденциальной информации и обязуюсь:
1. Не разглашать информацию, полученную в рамках обсуждения и выполнения задач по проекту, обеспечивать сохранность указанной информации и использование исключительно в целях реализации Проекта.
В контексте настоящего Обязательства понятие "Конфиденциальная информация" в рамках реализации Проекта означает любую информацию, сообщенную устно либо представленную в письменной, визуальной формах, на электронных носителях или иной материальной форме, включая, но, не ограничиваясь перечисленной в настоящем Обязательстве информацией:
- персональные данные граждан (пациентов), работников НАЗВАНИЕ ОРГАНИЗАЦИИ и ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, третьих лиц;
- сведения, составляющие врачебную тайну, в т.ч. информация о состоянии здоровья граждан (пациентов), диагноз, иные сведения, полученные при обследовании и лечении граждан (пациентов);
- документация и сведения по Проекту, в т.ч. почтовые письма и служебная переписка;
- программное обеспечение, аппаратные и программных средства, используемые для реализации Проекта;
- доступы (логины и пароли) и учетные записи;
- информация об иных проектах и деятельности НАЗВАНИЕ ОРГАНИЗАЦИИ и ФБУН ЦНИИ Эпидемиологии Роспотребнадзора, связанной с реализацией вышеуказанных нормативных актов (если данная информация не является публичной и не находится в открытом доступе).
2. Не передавать Конфиденциальную информацию лицам, не являющимся участниками Проекта, за исключением директора НАЗВАНИЕ ОРГАНИЗАЦИИ, ФБУН ЦНИИ Эпидемиологии Роспотребнадзора или иного специально уполномоченного Правительством Российской Федерации или Роспотребнадзором по данному Проекту лица/организации;
3. Не копировать и не распространять Конфиденциальную информацию любым способом, кроме согласованного директором НАЗВАНИЕ ОРГАНИЗАЦИИ и ФБУН ЦНИИ Эпидемиологии Роспотребнадзора или иным специально уполномоченным Правительством РФ или Роспотребнадзором по данному Проекту лицом/организацией;
4. Ни во время моей работы, ни в течение 3 (трех) лет после увольнения не обсуждать и не раскрывать информацию, связанную как с деятельностью НАЗВАНИЕ ОРГАНИЗАЦИИ и ФБУН ЦНИИ Эпидемиологии Роспотребнадзора при реализации Проекта, так и информацию, касающиеся лично моего участия в Проекте (характер выполняемой работы, ее содержание и др.);
_____________________ _______________________________________________
(подпись) (фамилия, инициалы)
5. Обеспечить сохранность документов, содержащих Конфиденциальную информацию, не допускать несанкционированное ознакомление с документами, содержащими Конфиденциальную информацию, третьих лиц и работников НАЗВАНИЕ ОРГАНИЗАЦИИ, не привлеченных к работе по Проекту директором НАЗВАНИЕ ОРГАНИЗАЦИИ или иным специально уполномоченным Правительством РФ или Роспотребнадзором по данному Проекту лицом/организацией;
6. В случае попытки посторонних лиц получить от меня сведения, составляющие Конфиденциальную информацию, немедленно сообщать об этом директору НАЗВАНИЕ ОРГАНИЗАЦИИ или иному уполномоченному директором по данному Проекту лицу;
7. Об утрате или недостаче носителей Конфиденциальной информации и о других фактах, которые могут привести к разглашению Конфиденциальной информации, а также о причинах и условиях возможной утечки сведений и хищений немедленно сообщать директору НАЗВАНИЕ ОРГАНИЗАЦИИ или иному уполномоченному директором по данному Проекту лицу;
8. В случае моего увольнения вернуть все носители Конфиденциальной информации (документы, черновики, чертежи, зарисовки, макеты, магнитные ленты, диски, внешние накопители, распечатки на принтерах, фотонегативы и позитивы, модели, материалы, изделия, коды и пр.), которые находились в моем распоряжении в связи с выполнением мною должностных обязанностей во время работы над Проектом;
9. В случае вынужденного раскрытия любой Конфиденциальной информации или совершения любого другого действия в нарушение настоящего Обязательства, обязуюсь незамедлительно уведомить об этом директора НАЗВАНИЕ ОРГАНИЗАЦИИ в письменной форме;
10. Соблюдать иные требования, предусмотренные локальными нормативными актами ФБУН ЦНИИ Эпидемиологии Роспотребнадзора об обеспечении сохранности коммерческой тайны и конфиденциальной информации ФБУН ЦНИИ Эпидемиологии Роспотребнадзора.
11. Обеспечить незамедлительный возврат всех материалов, содержащих конфиденциальную информацию, а также всех копий таких материалов в дирекцию НАЗВАНИЕ ОРГАНИЗАЦИИ или иному специально уполномоченному им по данному Проекту лица.
Я не буду сам(а) и не позволю другим лицам без разрешения директора НАЗВАНИЕ ОРГАНИЗАЦИИ делать копии либо аннотации на документы и материалы, касающиеся Конфиденциальной информации и реализации Проекта.
Мне известно, что нарушение настоящего Обязательства может повлечь дисциплинарную, административную, гражданско-правовую или уголовную ответственность в соответствии с законодательством Российской Федерации: в виде дисциплинарного взыскания, вплоть до расторжения Трудового договора; обязанности по возмещению ущерба - убытков, упущенной выгоды и ущерба деловой репутации. Настоящее соглашение не распространяется на сведения, полученные мной до факта загрузки их в систему Проекта, и не ограничивает меня в плане работы с этими сведениями, либо их публикации.
_____________________ _______________________________________________
(подпись) (фамилия, имя, отчество)
"__" _________ 202___ года"
Приложение 4
к MP 3.1.0272-22
Перечень идентифицируемых генетических вариантов SARS-CoV-2
В настоящее время (на 10.01.2022) к вариантам ВЭП (VOC) относятся штаммы, принадлежащие к линиям:
- Alpha, В.1.1.7 ("Британский");
- Beta, В.1.351 ("ЮАР");
- Gamma, P.1 линии В.1.1.28 ("Бразильский");
- Карра, В.1.617.1 ("Индийский. 1");
- Delta, В.1.617.2 ("Индийский.2").
- Omicron, В.1.1.529 ("Южно-Африканский")
К вариантам ТДИ (VUI) (на 12.06.2021) относятся:
- АТ.1 линии В.1.1.370.1;
- В.1.1.523 (ранее относился к линии В.1.1.451, согласно PANGOILIN)
Ниже приводятся рекомендации по дифференциации вышеуказанных геновариантов с помощью методов (1) фрагментного секвенирования по Сэнгеру отдельных локусов гена, кодирующего S-белок, (2) секвенирования по Сэнгеру полного гена S-белка, (3) полногеномного секвенирования.
Информация о характерных мутациях S-гена для каждой линии приведена в таблице 1.
Таблица 1. Характерные мутации S-гена геновариантов ВЭП и ТДИ
| Мутации гена S | |
|---|---|
| Alpha, В.1.1.7 | Н69-, V70-, Y144-, N501Y, A570D, D614G, Р681Н, T716I, S982A, D1118H |
| Beta, В.1.351 | D80A, D215G, L241-, L242-, А243-, K417N, Е484К, N501Y, D614G, A701V |
| Gamma, P.1 | L18F, T20N, P26S, D138Y, R190S, К417Т, Е484К, N501Y, D614G, H655Y, T1027I, V1176F |
| Delta, В.1.617.2 | T19R, Е156-, F157-, R158G, L452R, Т478К, D614G, P681R, D950N |
| Карра, В.1.617.1 | G142D, Е154К, L452R, E484Q, D614G, P681R, Q1071H |
| AT. 1 линии В.1.1.370.1 | P9L, D215G, C136-, N137-, D138-, Р139-, F140-, L141-, G142-, V143-, Y144-, Н245Р, F306I, T307S, V308C, E309R, K310S, G311V, I312L, Е484К, N679K, ins_679_GIAL, E780K |
| В.1.1.523 | E156-, F157-, R158-, F306L, E484K, S494P, E780A |
| В.1.1.529 |
1. Алгоритм дифференциации генетических вариантов вируса SARS-CoV-2 с помощью фрагментного секвенирования по Сэнгеру отдельных локусов гена, кодирующего S-белок.
Дифференциацию геновариантов вируса SARS-CoV-2 по локусам из протоколов Университета Женевы и ARTIC осуществляют в соответствии с таблицей 2, определяя делеции 21765-21770 (делеция HV 69-70), 21991-21993 (делеция Y144), 22287-22295 (делеция LAL 242-244), 21969-21995 (делеция CNDPFLGNY 136-144), замены А21801С (замена D80A), G21974T (замена D138Y), G22132T (замена R190S), A22206G (замена D215G), А22296С (замена Н245Р), T22917G (замена L452R), С22995А (замена Т478К), G23012A (замена Е484К), G23012C (замена E484Q), А23063Т (замена N501Y), С23271А (замена A570D). Нумерация нуклеотидных остатков приведена относительно референс-штамма hCoV-19/Wuhan/WIV04/2019 (EPI_JSL_402124).
На идентификацию геноварианта В.1.1.7 ("Британский") направлено обнаружение при секвенировании фрагмента F44-R44 или nCoV-2019_72 делеций 21765-21770 (HIV 69-70), 21991-21993 (Y144), а также обнаружение замены А23063Т (N501Y) при секвенировании фрагмента F47-R47 или nCoV-2019_76, замены С23271А (A570D) при секвенировании фрагмента F47-R47 или nCoV-2019_77.
На идентификацию геноварианта В.1.351 ("ЮАР") направлено обнаружение при секвенировании фрагмента F44-R44 или nCoV-2019_72 замены А21801С (D80A), а также обнаружение делеции 22287-22295 (LAL 242-244) и замены A22206G (D215G) при секвенировании фрагмента nCoV-2019_73, замен G23012A (Е484К) и А23063Т (N501Y) при секвенировании фрагмента F47-R47 или nCoV-2019_76.
На идентификацию геноварианта -Р.1 линии В.1.1.28 ("Бразильский") направлено обнаружение при секвенировании фрагмента F44-R44 или nCoV-2019_72, замены G21974T (D138Y), а также обнаружение замены G22132T (R190S) при секвенировании фрагмента nCoV-2019_73, замен G23012A (Е484К) и А23063Т (N501Y) при секвенировании фрагмента F47-R47 или nCoV-2019_76.
На идентификацию геноварианта В.1.617.1 ("Индийский.1") направлено обнаружение при секвенировании nCoV-2019_76 замен T22917G (L452R) и G23012C (E484Q) для филогенетической линии В.1.617.1 и замен T22917G (L452R).
На идентификацию геноварианта В.1.617.2 ("Индийский.2") направлено обнаружение при секвенировании nCoV-2019_76 замен T22917G (L452R) и G23012C (E484Q) для филогенетической линии В.1.617.1 и замен T22917G (L452R), а также С22995А (Т478К) для филогенетической линии В.1.617.2.
На идентификацию геноварианта В.1.1.523 направлено обнаружение при секвенировании фрагмента F44-R45 или nCoV-2019_(72_Left+73_Right) делеции 22029-22037 (EFR 156-158), замен G23012A (Е484К) и t23042c (S494P) при секвенировании фрагмента F47-R47i или nCoV-2019_76.
На идентификацию геноварианта АТ.1 линии В.1.1.370.1 направлено обнаружение при секвенировании фрагмента F44-R44 или nCoV-2019_72 делеции 21969-21995 (CNDPFLGNY 136-144), а также обнаружение замен A22206G (D215G), А22296С (Н245Р) при секвенировании фрагмента nCoV-2019_73, замены G23012A (Е484К) при секвенировании фрагмента F47-R47 или nCoV-2019_76.
Таблица 2. Список мутаций генетических вариантов вируса SARS-CoV-2, детектируемых при фрагментом секвенировании
| По протоколу Университета Женевы | По протоколу ARTIC v.3 | Генетические варианты SARS-CoV-2 | ||||
|---|---|---|---|---|---|---|
| Фрагмент F44-R45, внутренний фрагмент F44-R44*(1) | Фрагмент F46-R47, внутренний фрагмент F47-R47 | Фрагмент nCoV-2019_72 | Фрагмент nCoV-2019_73 | Фрагмент nCoV-2019_76 | Фрагмент nCoV-2019_77 | |
| Делеция HIV69-70, Y144 | Замены N501Y A570D | Делеция HIV69-70, Y144 | нет | Замена N501Y | Замена A570D | Alpha B.1.1.7 (Британский) |
| Замена D80A | Замены N501Y Е484К | Замена D80A | Делеция LAL 242-244 Замена D215G | Замены N501Y Е484К | нет | Beta В.1.351 (ЮАР) |
| Замена D138Y | Замены N501Y Е484К | Замена D138Y | Замена R190S | Замены N501Y Е484К | нет | Gamma P.1, B.1.1.28 (Бразилия) |
| Замены G142D*(2) Е154К*(2) | Замены L452R*(3) E484Q | Замена Т951*(2) G142D | нет | Замены L452R E484Q | нет | Карра В.1.617.1 (Индия.1) |
| Делеция FR156-157*(1) Замена R158G*(1) | Замены L452R*(3) Т478К*(3) | нет | Делеция FR156-157*(1) Замена R158G*(1) | Замены L452R Т478К | нет | Delta В.1.617.2 (Индия.2)*(1) |
| Делеция EFR156-158*(1) | Е484К S494P | нет | Делеция EFR156-158 | Е484К S494P | нет | В.1.1.523 |
| Делеция CNDPFLGNY136-144 | Е484К*(4) | Делеция CNDPFLGNY 136-144 | Замена*(5) D215G Н245Р | Е484К | нет | АТ.1 линии В.1.1.370.1 (северо-западный)*(4), *(5) |
______________________________
*(1) Использование праймера R44 из протокола Университета Женевы и nCoV-2019_72_Right из протокола ARTIC v.3 может быть неэффективным для идентификации геноварианта В. 1.617.2 (Индия.2)! (праймеры относятся к зоне делеции).
*(2) Наличие данных замен и/или делеций является возможным, но не определяющим. Определяющими стоит считать замены в положениях 452, 478, 484.
*(3) Идентификация данных замен возможна только при секвенировании фрагмента F46-R47.
*(4) В случае геноварианта АТ.1 линии В.1.1.370.1 (северо-западный) использование праймера F46 из протокола Университета Женевы может быть неэффективным!
*(5) При определении геноварианта АТ.1 линии В.1.1.370.1 (северо-западный) использование праймера nCoV-2019_73_Left из протокола ARTIC v.3 невозможно!
Рекомендуемые протоколы исследования с помощью фрагментного секвенирования по Сэнгеру отдельных локусов гена, кодирующего S-белок:
Для амплификации участков, содержащих нуклеотидные замены, необходимые для определения геновариантов вируса SARS-CoV-2, используют олигонуклеотидные праймеры, указанные в Протоколе Университета Женевы (Geneva, December 26th, 2020, Rue Gabrielle-Perret-Gentil 4, 1211 Geneva 14, Switzerland), см. таблицу З.
Таблица 3. Праймеры используемые по протоколу Университета Женевы
| Обозначение | Последовательность (5′-3′) |
|---|---|
| F44 | TCTCTTCTTAGTAAAGGTAGACTT |
| F46* | CCTTCACTGTAGAAAAAGGAATC |
| F47 | TATCAGGCCGGTAGCACAC |
| R45 | CTAACAATAGATTCTGTTGGTTG |
| R44** | GAATAAACTCTGAACTCACTTTCC |
| R47 | CATATGAGTTGTTGACATGTTCAG |
______________________________
* В случае геноварианта АТ.1 линии В.1.1.370.1 (северо-западный) использование праймера F46 из протокола Университета Женевы может быть неэффективным!
** Использование праймера R44 из протокола Университета Женевы может быть неэффективным для идентификации геноварианта В.1.617.2 (Индия.2)! (праймеры попадают в зону делеции).
При исследовании проб с низкими значениями 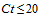 (высокая вирусная нагрузка) достаточно одного раунда ПЦР с праймерами F44-R44 и F47-R47, обеспечивающими образование фрагментов 570 п.н. и 565 п.н., соответственно.
(высокая вирусная нагрузка) достаточно одного раунда ПЦР с праймерами F44-R44 и F47-R47, обеспечивающими образование фрагментов 570 п.н. и 565 п.н., соответственно.
При исследовании проб с высокими значениями  (низкая вирусная нагрузка) используется гнездовая ПЦР.
(низкая вирусная нагрузка) используется гнездовая ПЦР.
Первоначально проводят амплификацию с праймерами F44-R45 и F46-R47, фланкирующими фрагменты размером 1071 п.н. и 1068 п.н., а затем осуществляют вторую ПЦР с праймерами F44-R44 и F47-R47, где в качестве исследуемой ДНК используют ампликоны, полученные в первом раунде, см. таблицу 4.
Таблица 4. Рекомендуемые условия проведения реакции амплификации при использовании Протокола Университета Женевы.
| Матричный режим | 1 этап | 2 этап | 5 этап | 6 этап |
|---|---|---|---|---|
| 95°С - 5 мин | 95°С - 30 с 55°С - 30 с 72°С - 70 с 38 циклов | 72°С - 4 мин | 10°С - хранение |
В качестве альтернативного способа рекомендуется использовать пары праймеров nCoV-2019_72, nCoV-2019_76 и nCoV-2019_77 из протокола ARTIC v.3 (nCoV-2019 sequencing protocol v3 (LoCost) (protocols.io), см. таблицу 5.
Таблица 5. Праймеры используемые по протоколу ARTIC v.3
| Обозначение | Последовательность (5′-3′) |
|---|---|
| nCoV-2019_72_LEFT | ACACGTGGTGTTTATTACCCTGAC |
| nCoV-2019_72_RIGHT | ACTCTGAACTCACTTTCCATCCAAC |
| nCoV-2019_73_LEFT | CAATTTTGTAATGATCCATTTTTGGGTGT |
| nCoV-2019_73_RIGHT | CACCAGCTGTCCAACCTGAAGA |
| nCoV-2019_76_LEFT | AGGGCAAACTGGAAAGATTGCT |
| nCoV-2019_76_RIGHT | ACACCTGTGCCTGTTAAACCAT |
| nCoV-2019_76_LEFT_alt3 | GGGCAAACTGGAAAGATTGCTGA |
| nCoV-2019_76_RIGHT_alt0 | ACCTGTGCCTGTTAAACCATTGA |
| nCoV-2019_77_LEFT | CCAGCAACTGTTTGTGGACCTA |
Рекомендуемые условия проведения реакции амплификации при использовании Протокола ARTIC v.3 см. таблица 6.
Таблица 6. Рекомендуемые условия проведения реакции амплификации при использовании протокола ARTIC v.3
| Матричный режим | 1 этап | 3 этап | 4 этап | 5 этап | 6 этап |
|---|---|---|---|---|---|
| 95°С - 5 мин | 95°С - 60 с 62°С - 60 с 72°С - 60 с 10 циклов | 95°С - 30 с 60°С - 30 с 72°С - 30 с 30 циклов | 72°С - 5 мин | 10°С - хранение |
Допустимо также использовать праймеры из иных протоколов, позволяющие проводить амплификацию и последующее фрагментное секвенирование участков генома SARS-CoV-2, содержащих перечисленные нуклеотидные замены.
В качестве замены праймерам R44 из протокола Университета Женевы и nCoV-2019_72_Right из протокола ARTIC v.3 может быть использован праймер CACV_51_R, праймеру F46 из протокола Университета Женевы - праймер CACV_55_F и праймеру nCoV-2019_73_Left из протокола ARTIC v.3 - праймер CACV_52_F (таблица 7).
Таблица 7. Праймеры, рекомендуемые для замены неработающих праймеров из протоколов Университета Женевы и протокола ARTIC v.3
| Название | Последовательность 5′-3′ | Длина, пн |
|---|---|---|
| CACV_51_R | GAG GGA GAT CAC GCA СТА АА | 20 |
| CACV_55_F | ATG GAA CCA TTA CAG ATG CTG TAG | 24 |
| CACV_52_F | TGG ATG GAA AGT GAG TTC AGA G | 22 |
2. Алгоритм дифференциации генетических вариантов вируса SARS-CoV-2 с помощью фрагментного секвенирования полной последовательности гена, кодирующего S-белок.
Для дифференциации генетических вариантов вируса SARS-CoV-2 с помощью фрагментного секвенирования полной последовательности гена, кодирующего S-белок предлагается использовать тот же алгоритм, что и указанный выше для фрагментного секвенирования отдельных локусов.
Для амплификации полного гена S, необходимого для определения геновариантов вируса SARS-CoV-2, используют олигонуклеотидные праймеры из протокола ARTIC v3, перекрывающие полную нуклеотидную последовательность гена S SARS-CoV-2 длиной 3822 нуклеотидных оснований (позиция в геноме 21562 - 25384 н.о.).
Их последовательности и позиции в геноме, а также длина ожидаемых ампликонов, указаны ниже (см. таблица 8). Рекомендуемые условия проведения реакции амплификации при использовании протокола ARTIC v.3 приведены в таблице 8.
Таблица 8. Нуклеотидные последовательности и позиции в геноме олигонуклеотидных праймеров для секвенирования гена S SARS-CoV-2 (протокол ARTIC v3).
* ВНИМАНИЕ! Структура праймера N 1 (nCoV-2019_72_LEFT_alt) является уникальной, не входит в набор оригинального протокола ARTIC v3 и требует отдельного заказа.
| name | Позиция | seq | length | %gc | Длина ампликона | |
|---|---|---|---|---|---|---|
| 1 | nCoV-2019_72_LEFT_alt | 21508-21544 | FGAGTTGTTATTTCTAGTGATGTTCTTG | 27 | 33.00 | 530 |
| 2 | nCoV-2019_72_RIGHT | 22014-22038 | ACTCTGAACTCACTTTCCATCCAAC | 25 | 44.00 | |
| 3 | nCoV-2019_73_LEFT | 21962-21990 | CAATTTTGTAATGATCCATTTTTGGGTGT | 29 | 31.03 | 384 |
| 4 | nCoV-2019_73_RIGHT | 22327-22346 | CACCAGCTGTCCAACCTGAAGA | 22 | 54.55 | |
| 5 | nCoV-2019_74_LEFT | 22263-22290 | ACATCACTAGGTTTCAAACTTTACTTGC | 28 | 35.71 | 387 |
| 6 | nCoV-2019_74_RIGHT | 22627-22650 | GCAACACAGTTGCTGATTCTCTTC | 24 | 45.83 | |
| 7 | nCoV-2019_75_LEFT | 22517-22542 | AGAGTCCAACCAACAGAATCTATTGT | 26 | 38.46 | 386 |
| 8 | nCoV-2019_75_RIGHT | 22878-22903 | ACCACCAACCTTAGAATCAAGATTGT | 26 | 38.46 | |
| 9 | nCoV-2019_76_LEFT_alt3 | 22799-22821 | GGGCAAACTGGAAAGATTGCTGA | 23 | 47.83 | 413 |
| 10 | nCoV-2019_76_RIGHT_alt0 | 23190-23212 | ACCTGTGCCTGTTAAACCATTGA | 23 | 43.48 | |
| 11 | nCoV-2019_77_LEFT | 23123-23144 | CCAGCAACTGTTTGTGGACCTA | 22 | 50.00 | 399 |
| 12 | nCoV-2019_77_RIGHT | 23501-23522 | CAGCCCCTATTAAACAGCCTGC | 22 | 54.55 | |
| 13 | nCoV-2019_78_LEFT | 23444-23466 | CAACTTACTCCTACTTGGCGTGT | 23 | 47.83 | 403 |
| 14 | nCoV-2019_78_RIGHT | 23823-23847 | TGTGTACAAAAACTGCCATATTGCA | 25 | 36.00 | |
| 15 | nCoV-2019_79_LEFT | 23790-23812 | GTGGTGATTCAACTGAATGCAGC | 23 | 47.83 | 379 |
| 16 | nCoV-2019_79_RIGHT | 24146-24169 | CATTTCATCTGTGAGCAAAGGTGG | 24 | 45.83 | |
| 17 | nCoV-2019_80_LEFT | 24079-24100 | TTGCCTTGGTGATATTGCTGCT | 22 | 45.45 | 388 |
| 18 | nCoV-2019_80_RIGHT | 24444-24467 | TGGAGCTAAGTTGTTTAACAAGCG | 24 | 41.67 | |
| 19 | nCoV-2019_81_LEFT | 24292-24416 | GCACTTGGAAAACTTCAAGATGTGG | 25 | 44.00 | 497 |
| 20 | nCoV-2019_81_RIGHT | 24766-24789 | GTGAAGTTCTTTTCTTGTGCAGGG | 24 | 45.83 | |
| 21 | nCoV-2019_82_LEFT | 24697-24721 | GGGCTATCATCTTATGTCCTTCCCT | 25 | 48.00 | 379 |
| 22 | nCoV-2019_82_RIGHT | 25053-25076 | TGCCAGAGATGTCACCTAAATCAA | 24 | 41.67 | |
| 23 | nCoV-2019_83_LEFT | 24979-25003 | TCCTTTGCAACCTGAATTAGACTCA | 25 | 40.00 | 390 |
| 24 | nCoV-2019_83_RIGHT | 25401-25369 | TTTGACTCCTTTGAGCACTGGC | 22 | 50.00 |
Таблица 9. Рекомендуемые условия проведения реакции амплификации при использовании праймеров из протокола ARTIC v3.*
| Матричный режим | 1 этап | 3 этап* | 5 этап | 6 этап |
|---|---|---|---|---|
| 95°С - 30 с | 95°С - 30 с 55°С - 10 с 72°С - 40 с 30-35 циклов | 72°С - 2 мин | 10°С - хранение |
______________________________
* данный температурный режим указан для протокола на основе Q5® High-Fidelity DNA Polymerase. При использовании других ферментов может потребоваться проведение оптимизации условий реакции.
При невозможности использования праймеров из протокола ARTIC v.3 предлагается использовать альтернативный набор праймеров из протокола ЦНИИЭ (неопубликованные данные) или отдельные праймеры, см. таблица 10.
Таблица 10. Нуклеотидные последовательности и позиции в геноме олигонуклеотидных праймеров для секвенирования гена S SARS-CoV-2 (протокол ЦНИИЭ)
| Название праймера | Позиция | Последовательность 5′-3′ | Длина | Длина ампликона, пн |
|---|---|---|---|---|
| Cacv51_F | 21420-21440 | AAG GGG TAC TGC TGT TAT GTC | 21 | 795 |
| Cacv51_R | 22195-22214 | GAG GGA GAT CAC GCA СТА АА | 20 | |
| Cacv52_F | 22016-22037 | TGG ATG GAA AGT GAG TTC AGA G | 22 | 526 |
| Cacv52_R | 22518-22541 | CAA TAG ATT CTG TTG GTT GGA CTC | 24 | |
| Cacv55_F | 22407-22430 | ATG GAA CCA TTA СAG ATG CTG TAG | 24 | 585 |
| Cacv55_R | 22971-22991 | TAC CGG CCT GAT AGA TTT CAG | 21 | |
| Cacv07_F | 22842-22861 | ATG ATT TTA CAG GCT GCG TT | 20 | 637 |
| Cacv08_R | 23072-23091 | CCT GTA GAA TAA АСА CGC CA | 20 | |
| Cacv81_F | 23261-23282 | AGA GAC ATT GCT GAC ACT ACT G | 22 | 585 |
| Cacv81_R | 23821-23845 | TGT АСА AAA ACT GCC ATA TTG CAA С | 25 | |
| Cacv82_F | 23684-23709 | TCT AAT AAC TCT ATT GCC ATA CCC AC | 26 | 538 |
| Cacv82_R | 24200-24221 | TCC AAC CAG AAG TGA TTG TAC С | 22 | |
| Cacv83_F | 24122-24145 | AAG TTT AAC GGC CTT ACT GTT TTG | 24 | 592 |
| Cacv83_R | 24690-24713 | АСА TAA GAT GAT AGC CCT TTC CAC | 24 | |
| Cacv84_F | 24587-24611 | ACT CAA CAA TTA ATT AGA GCT GCA G | 25 | 585 |
| Cacv84_R | 25149-25171 | AAG TTC TTG GAG АТС GAT GAG AG | 23 | |
| Cacv85_F | 24810-24833 | ATG ATG GAA AAG CAC ACT TTC CTC | 24 | 666 |
| Cacv09_R | 25281-25300 | AAA TCT GAA GGA GTA GCA TCC TTG | 24 |
Рекомендуемые условия проведения реакции амплификации при использовании праймеров из протокола ЦНИИЭ приведены в таблице 11.
Таблица 11. Рекомендуемые условия проведения реакции амплификации при использовании праймеров из протокола ЦНИИЭ
| Матричный режим | 1 этап | 3 этап* | 5 этап | 6 этап |
|---|---|---|---|---|
| 95°С - 5 мин | 95°С - 30 с 52°С - 30 с 72°С - 60 с 30-35 циклов | 72°С - 5 мин | 10°С - хранение |
______________________________
* данный температурный режим указан для протокола на основе Taq-полимеразы. При использовании других ферментов может потребоваться проведение оптимизации условий реакции.
Допустимо также использовать праймеры из иных протоколов, позволяющие проводить амплификацию и последующее фрагментное секвенирование последовательности S-гена SARS-CoV-2.
3. Алгоритм дифференциации генетических вариантов вируса SARS-CoV-2 с помощью проведения полногеномного секвенирования
Для заключения о геноварианте SARS-CoV-2 в исследованной пробе рекомендуется использовать алгоритм классификации PANGOLIN (https://cov-lineages.org/).
Для секвенирования полной последовательности геномов SARS-CoV-2 рекомендуется использовать (на выбор) праймерную панель и протокол подготовки библиотек:
- ARTIC v.3 https://www.protocols.io/view/ncov-2019-sequencing-protocol-bbmuik6w
- SCV-2000bp https://www.protocols.io/view/protocol-for-scv-2000bp-a-primer-panel-for-sars-co-bn77mhrn.html
- любые иные праймерные панели и протоколы, позволяющие осуществить амплификацию полного генома SARS-CoV-2".
Приложение 5
к MP 3.1.0272-22
Инструкция по работе с программой genome.crie.ru
Интерфейс работы с образцами.
Версия 1.4.0.
Управление документом.
| Авторы | ФБУН ЦНИИ Эпидемиологии, Информационно-аналитический отдел. |
|---|---|
| Файл | |
| Создан | 06.02.2021 |
| Последнее редактирование | 03.03.2021 |
| Количество страниц | 11 |
| Версия | Дата изменения | Описание изменения | Автор изменения |
|---|---|---|---|
| 1.0.0 | 08.02.21 | Составлена первая версия инструкции | Евстифеев Е.А. |
| 1.1.0 | 10.02.21 | Обновлены скриншоты из программы | Евстифеев Е.А. |
| 1.1.0 | 10.02.21 | Добавлено дополнительное описание при заполнении формы единичной загрузки | Евстифеев Е.А. |
| 1.2.0 | 03.03.21 | Обновлено описание полей реестра. | Евстифеев Е.А. |
| 1.3.0 | 28.04.21 | Добавлено описание для работы региона, выделено в отдельную инструкцию. | Евстифеев Е.А. |
| 1.4.0 | 07.05.21 | Обновлено описание по меню | Евстифеев Е.А. |
Предназначение системы
Проект - Российская платформа агрегации информации о геноме.
Задача системы - обеспечение быстрого доступа к данным об эпидемиях и пандемических вирусах.
Реализация проекта направлена на быстрый обмен данными по вирусу, в частности, по коронавирусу, вызывающем COVID-19. Это включает генетическую последовательность и соответствующие клинические и эпидемиологические данные, связанные с вирусами человека, а также географические и иные данные, связанные с вирусами. Это позволяет исследователям понять, как вирусы развиваются и распространяются во время эпидемий и пандемий.
Предоставление доступа
Доступ представляет собой ссылку на адрес для входа, логин и пароль. Доступ предоставляется посредством электронного обмена.
Авторизация в системе
При входе в систему появляется окно с запросом на авторизацию. В окне необходимо ввести логин и пароль, предоставленные пользователю, затем нажать кнопку "Войти":

Данное окно доступно только для авторизованных пользователей, обладающих правами по работе с образцами.

В нем доступен следующий функционал:
1. Новый образец
2. Образцы
3. Реестр образцов
3.1. Создать реестр
3.2. Список реестров
1. Новый образец
По нажатию на данный пункт открывается окно оформления нового образца:
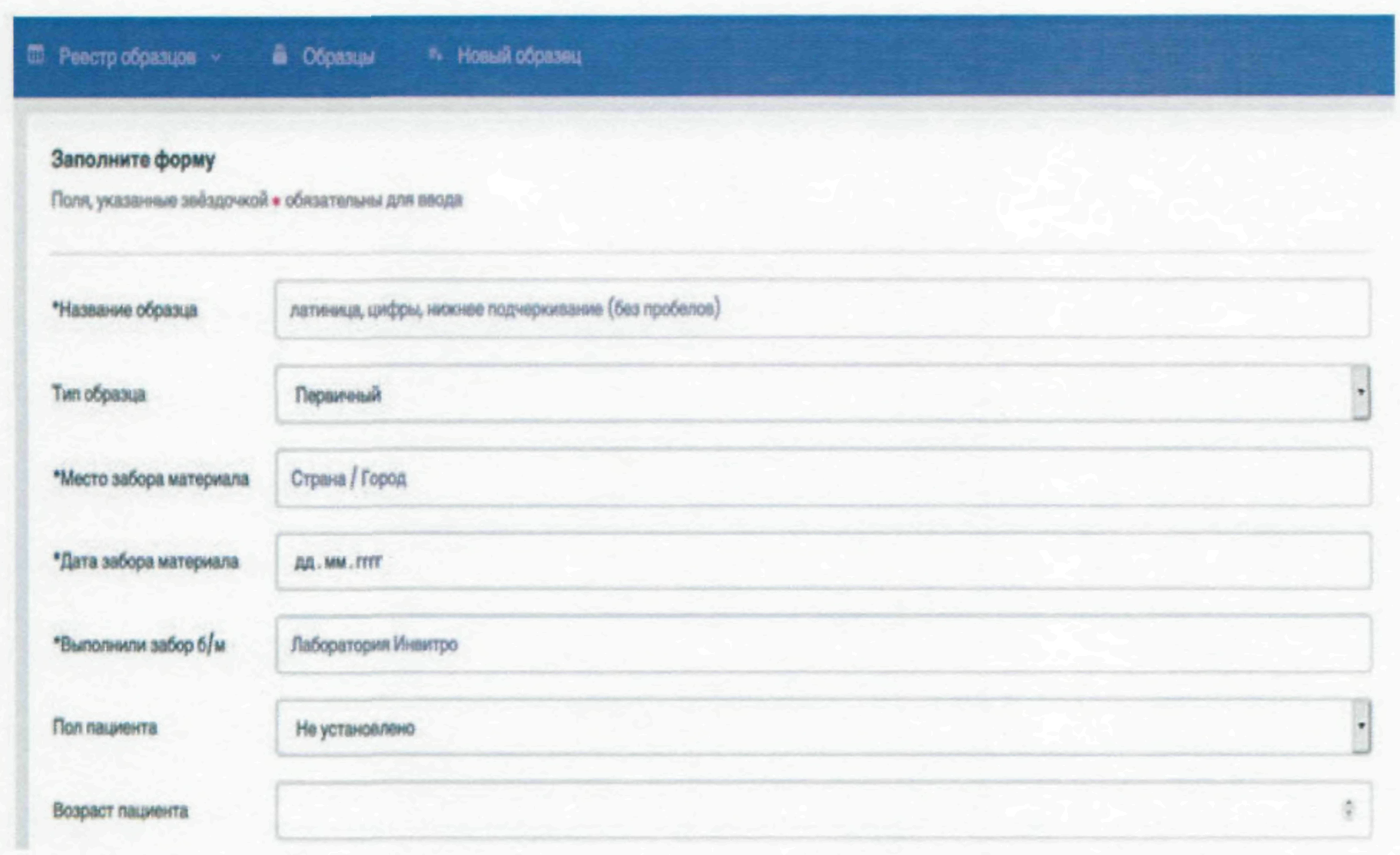
После загрузки файла и заполнения данных система отобразит окно об успешности загрузки:

Затем можно сразу начать оформление нового образца, либо посмотреть результат сохранения в разделе "Образцы".
2. Образцы
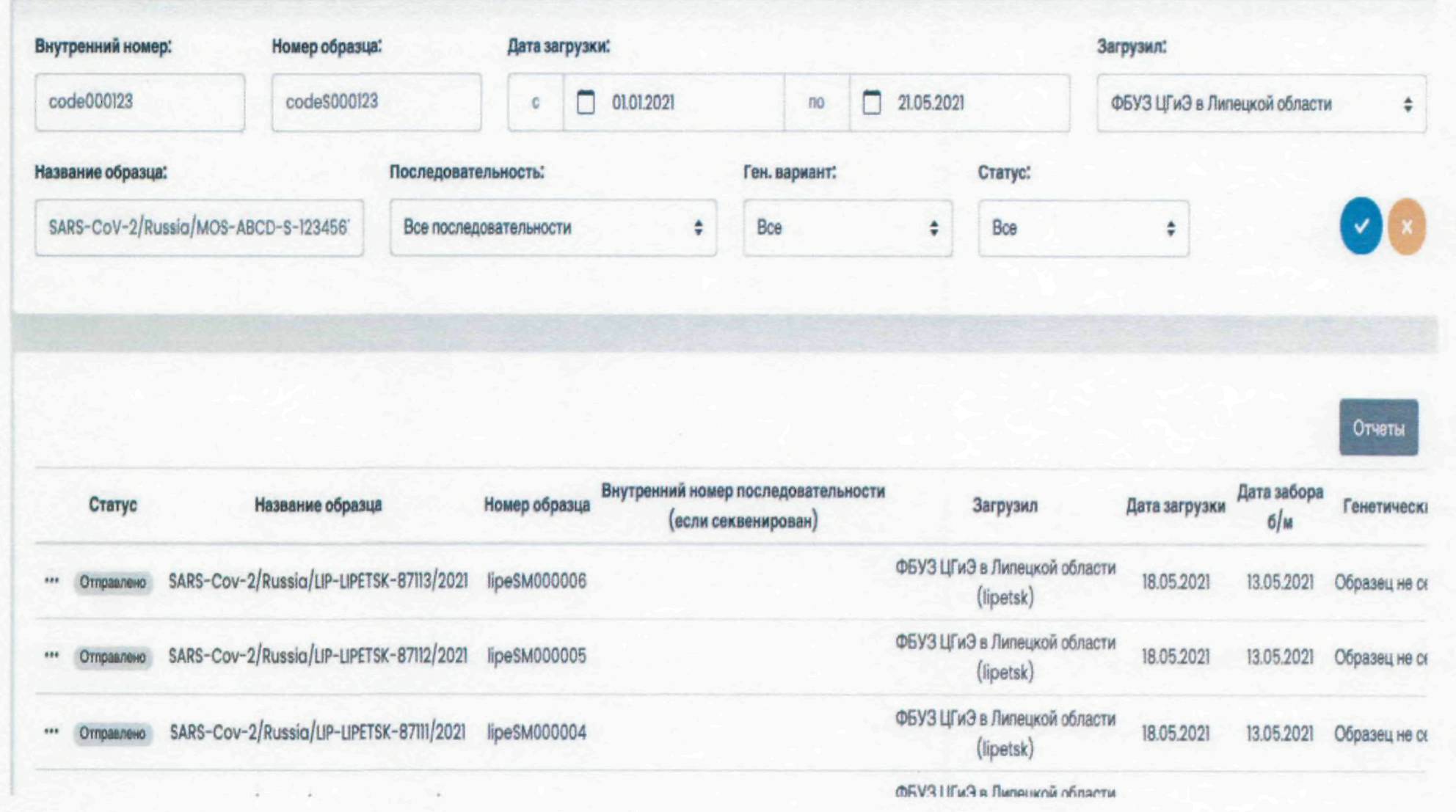
В данном разделе представлены все образцы с их статусами. По мере прохождения по процессу обработки образца статус будет изменяться.
В правом верхнем углу расположена кнопка "Отчеты". Она позволяет выгружать данные, которые отображены на экране, в виде отчета.

Таким образом, есть возможность сформировать отчет и получить результаты по всем образцам в статусе "Предварительный результат", либо только готовы образцы.
3. Реестр образцов
3.1. Создать реестр
Для создания реестра необходимо перейти в раздел Реестры -> Создать реестр.

В нем можно выбрать все образцы, доступные для отправки посредством создания реестра.
С помощью чек-бокса галочками выбираются нужные образцы:

После нажатия кнопки требуется подтвердить отправку реестра с данными записями:
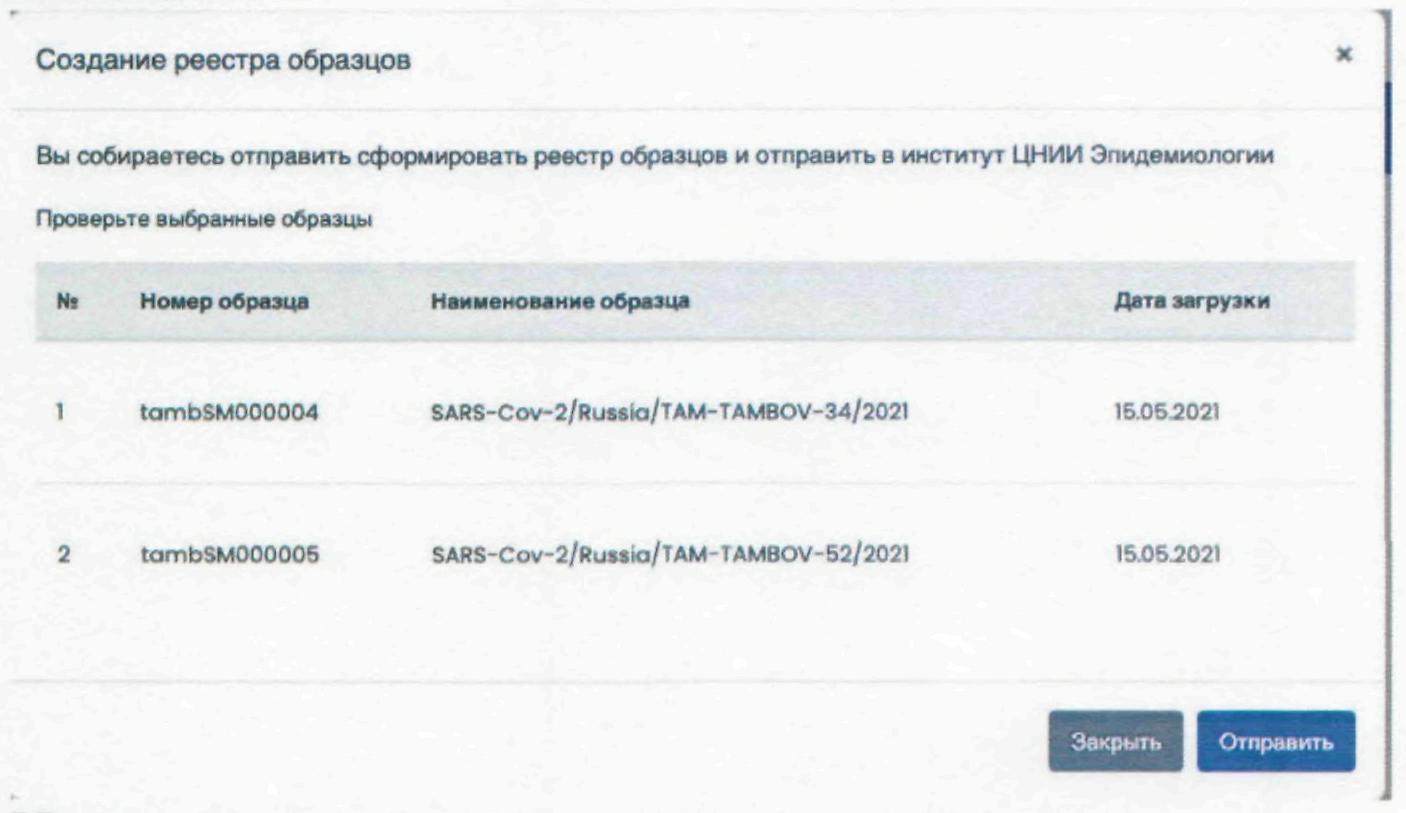
На следующем шаге распечатать сам реестр:
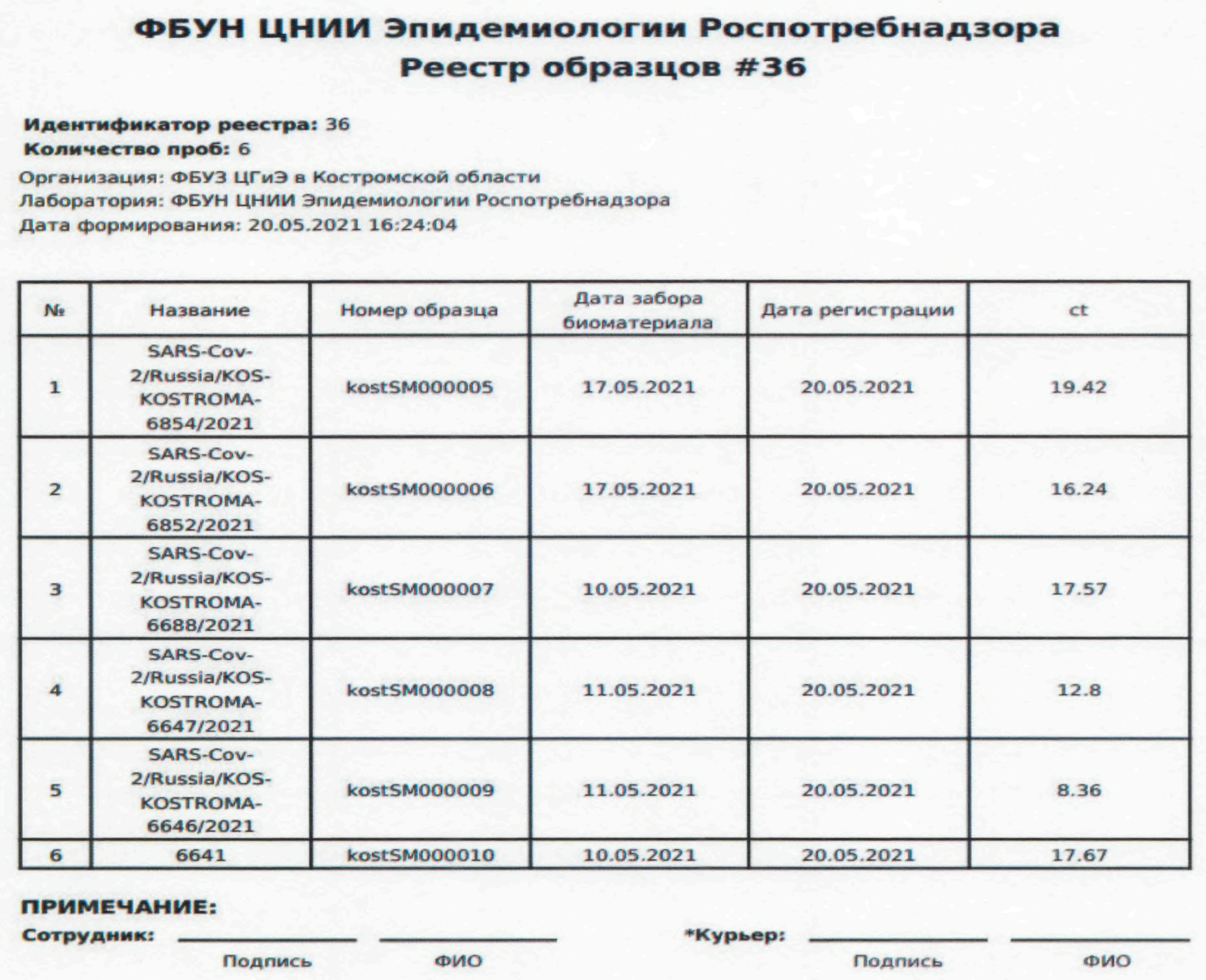
Полученный реестр нужно подписать и передать курьеру.
3.2. Список реестров
В данном разделе доступна история всех отправленных реестров. При нажатии на его номер можно открыть конкретный реестр и распечатать его повторно.
В самой таблице указана как дата отправки, так и дата принятия реестра.

Реестр может находится в 2 статусах:
- "Принят";
- "Отправлен".
|
Руководитель Федеральной службы по надзору в сфере защиты прав потребителей и благополучия человека, Главный государственный санитарный врач Российской Федерации |
А.Ю. Попова |
Обзор документа
Роспотребнадзор разработал рекомендации по молекулярно-генетическому мониторингу штаммов возбудителя коронавируса.

(1).jpg)


